乳腺癌是发生在乳腺上皮组织的恶性肿瘤,它是威胁全球女性健康的严重的常见的恶性肿瘤之一,近年来的发病率与死亡率也逐渐升高。因此,寻找和发现有价值的乳腺癌生物标志物已经成为目前研究的一个重要方向。小编这次为大家分享两篇关于研究乳腺癌 DNA 甲基化标志物的文章(IF > 10), 利用多组学数据分别从两个方向筛选甲基化标志物的研究。
-01-
案例一
易感基因 BRCA1 和 BCRA2 通过同源重组(Homologous Recombination,HR)对 DNA 进行修复,当 BRCA1 和 BCRA2 发生致病性突变,导致同源重组缺陷(HR-deficient,HRD),损伤的 DNA 难以得到修复,容易导致三阴性乳腺癌(TNBC)等恶性肿瘤的发生。40% - 70% 的 TNBC 肿瘤具有 HRD 表型,且 BRCA1/ BRCA2 突变的乳腺癌被认为比非 HR 缺陷瘤更具免疫原性。有研究表明,增加免疫原性可以起始更好的免疫检查点的连锁反应,但是这一机制仍不明确。瑞典隆德大学今年 7 月份在 Nat Commun(IF 12.121)上发表了《Comprehensive molecular comparison of BRCA1 hypermethylated and BRCA1 mutated triple negative breast cancers》, 通过全面比较 BRCA1 高甲基化和 BRCA1 突变的 TNBC 患者的分子生物学特征,以确定 BRCA1 甲基化水平在 TNBC 中的功能。

研究思路
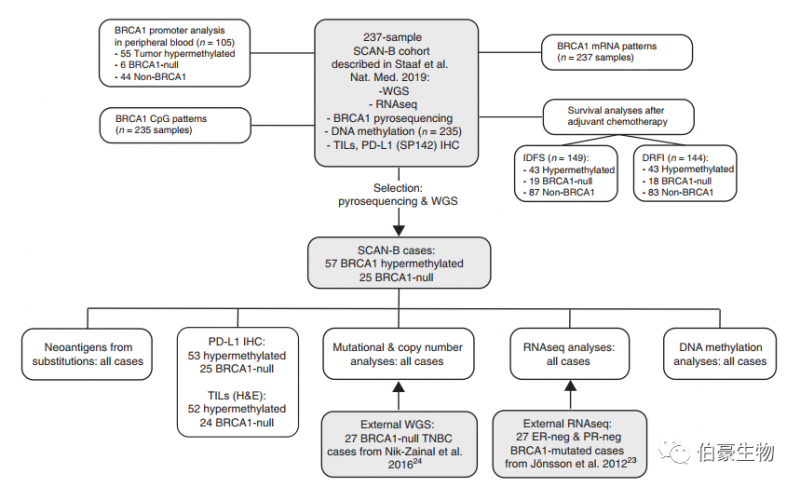
作者利用已经报道的 237 例未分群的三阴性乳腺癌(TNBCs)患者(SCAN-B)以及 54 例 BRCA1 突变肿瘤患者的 WGS、RNA-seq、WGBS、 焦磷酸测序数据, 分析了早期 TNBC 中 BRCA1 启动子高甲基化的频率,和 BRCA1 功能缺失(体细胞 / 种系突变、BRCA1 缺失)的肿瘤表型及其与临床病理变化、分子亚型和患者预后的关系。作者发现,在早期未分群的 TNBC 中,BRCA1 高甲基化的发生率是 BRCA1 功能缺失肿瘤的两倍,并且在肿瘤高甲基化患者的外周血 DNA 中可以检测到 BRCA1 启动子甲基化的升高。此外,从突变、表观遗传学、转录和免疫浸润表型分析中发现,BRCA1 高甲基化与 BRCA1 功能缺失患者的肿瘤表型几乎相同。与非 BRCA1 失活的 TNBC 患者相比,辅助化疗后 BRCA1 高甲基化和 BRCA1 功能缺失患者的预后更好, 说明 BRCA1 高甲基化很可能是早期 TNBC 发生的生物标志物。
结果展示
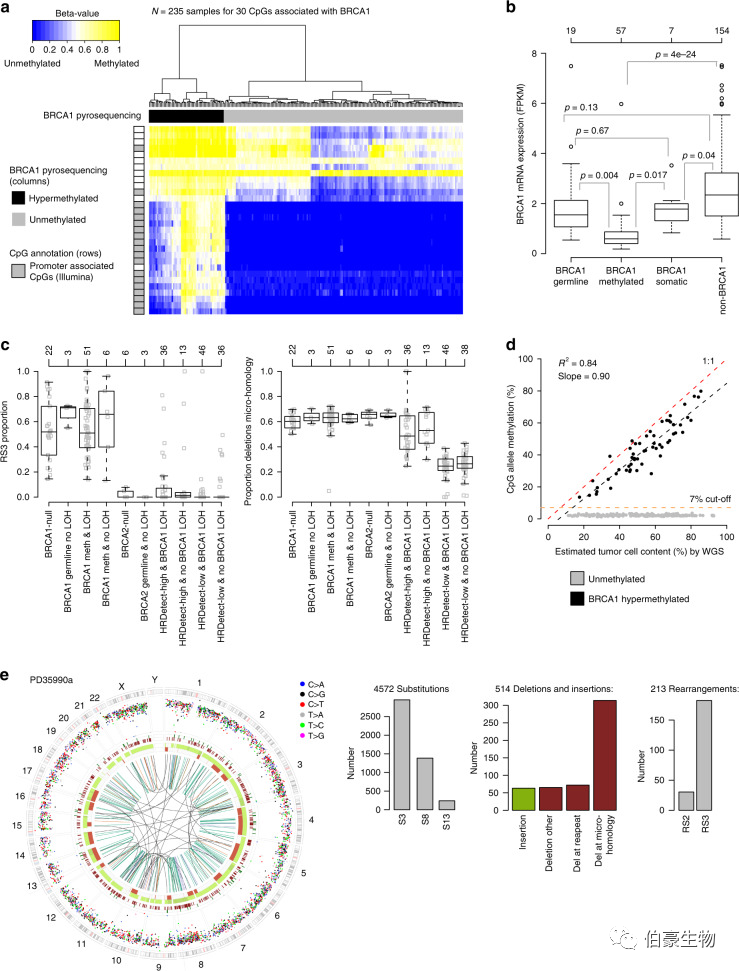
图 1. BRCA1 高甲基化,基因表达和 HRD 关联分析。作者通过对 235 例 TNBC 患者 BRCA1 启动子的焦磷酸测序结果和 237 例 TNBC 患者 BRCA1 mRNA 结果分析发现,高甲基化的 BRCA1 显著抑制了 mRNA 的表达。HRD 关联分析发现,BRCA1 缺陷癌症的典型特征是重排和微同源性缺失。且 WGS 结果表明,肿瘤细胞含量与 CpG 位点甲基化线性相关。这些结果显示,BRCA1 高甲基化比 BRCA1 缺失病例的发生频率高 2.3 倍,比 BRCA1 种系改变的发生频率高 3 倍。同时,作者对致病性的 BRCA2 双等位基因变异患者 PD35990a 进行分析发现,其肿瘤是以 BRCA1 缺陷为特征的 BRCA1 高甲基化增加和 BRCA1 LOH(loss of heterozygosity)等特点。
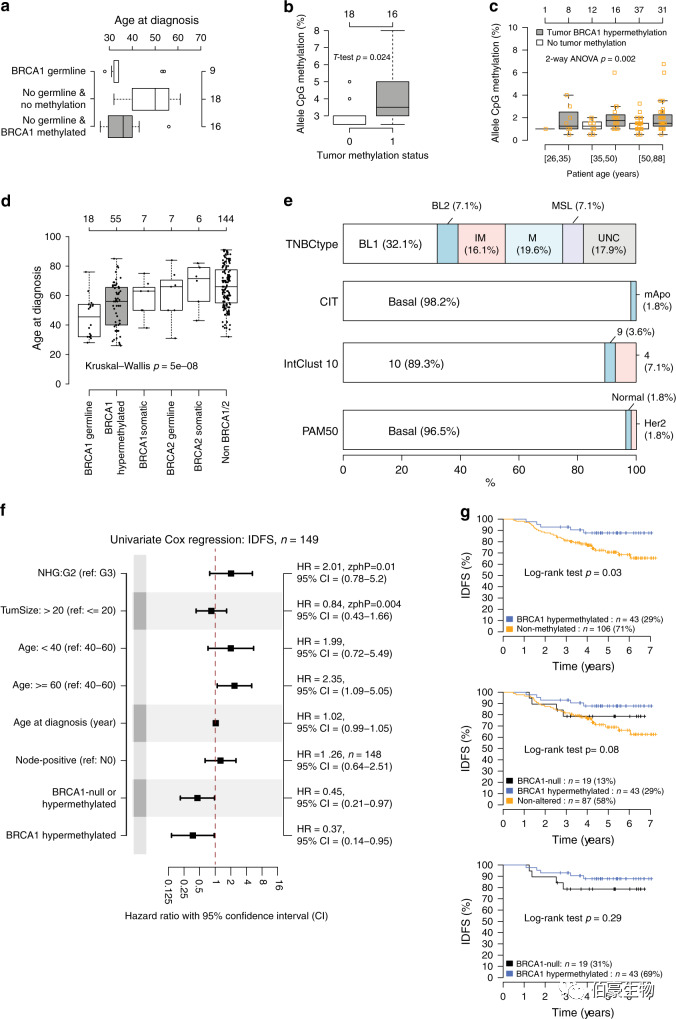
图 2. 外周血 BRCA1 甲基化、基因表达亚型和治疗后的预后。通过对 TNBC 患者年龄以及外周血甲基化分析显示,年轻患者(50 岁以下 TNBC 患者中 46.2%) 的 BRCA1 高甲基化频率较高,说明 BRCA1 高甲基化可能是 TNBC 患者的潜在致病因素。接着,作者用 Cox 分析和 KM 分析统计了 BRCA1 高甲基化患者和非 BRCA1 患者的生存情况,发现 BRCA1 高甲基化患者预后会更好。 说明 BRCA1 高甲基化患者预后改善的原因可能与 HRD 表型有关。
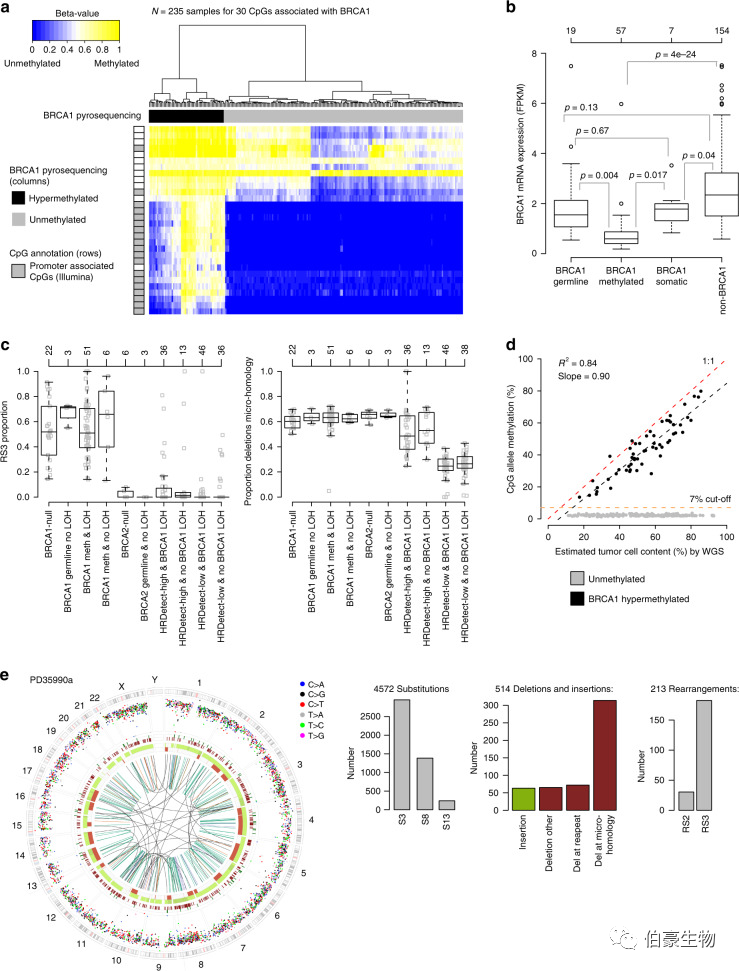
图 3. BRCA1 高甲基化和 BRCA1 缺失的 TNBC 患者的遗传表型比较。分别对 BRCA1 高甲基化和 BRCA1 缺失的 TNBC 患者的基因组拷贝数改变频率、驱动基因拷贝数扩增频率、驱动基因突变(插入、缺失、替换)的频率,两组样本的取代、indels 和重排总数以及包括两种 BRCA1/BRCA2 相关标记(RS3 和 RS5) 的六种重排标记和层次聚类、主成分分析进行比较,发现 BRCA1 高甲基化和 BRCA1 缺失的 TNBC 患者分子生物学特征接近。
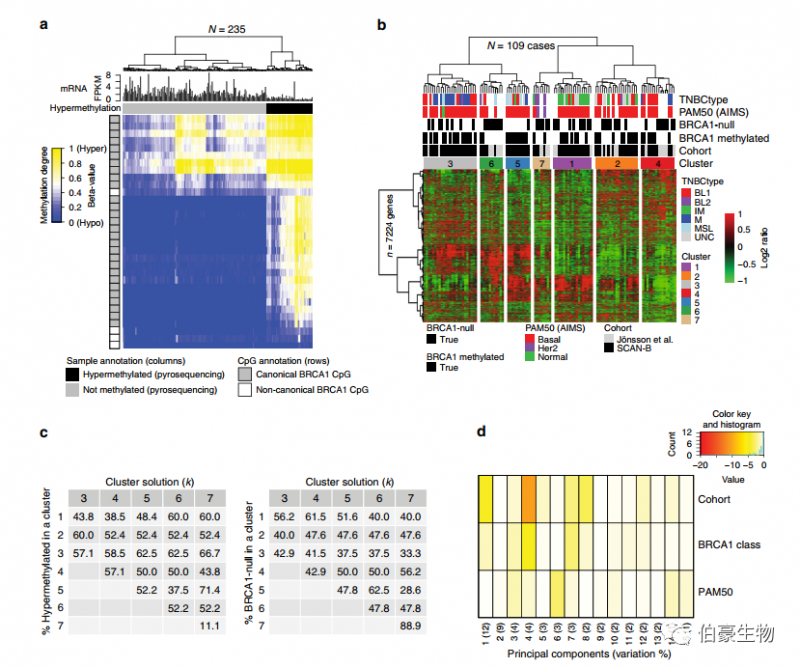
图 4 . BRCA1 高甲基化和 BRCA1 缺失的 TNBC 的 DNA 甲基化和转录特征。25 例 BRCA1 缺失和 57 例高甲基化 SCAN- B 病例的甲基化数据进行了比较,差异甲基化分析确定了 32 个显著的 CpGs 且都与 BRCA1 相关。对 32 个 CpGs 的聚类分析发现在 235 个 TNBC 患者中,BRCA1 高甲基化和非甲基化的 TNBC 有明显差异。转录组无监督聚类和主成分分析显示,BRCA1 高甲基化和 BRCA1 缺失的 TNBC 患者的基因表达无明显改变。
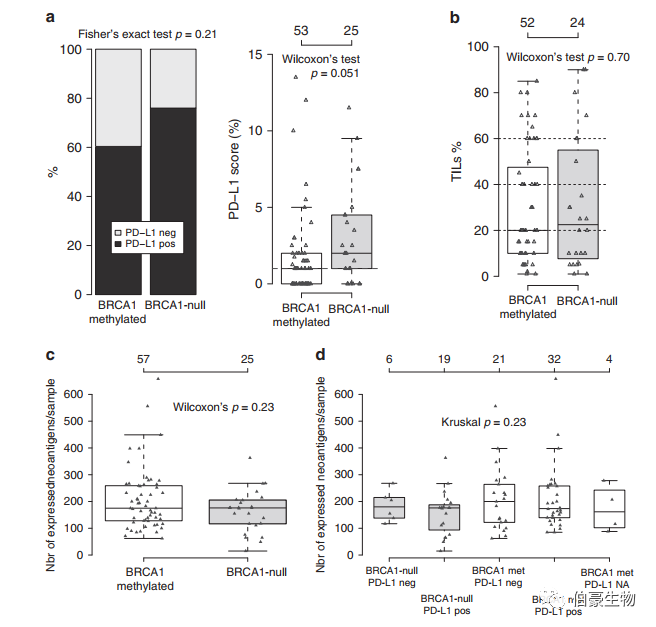
图 5. BRCA1 高甲基化和 BRCA1 缺失的免疫细胞浸润表型。在免疫组化实验中评估 53 例 BRCA1 高甲基化和 25 例 BRCA1 缺失患者的 PD-L1 评分,结果显示无显著差异。
(https://doi.org/10.1038/s41467-020-17537-2)
-02-
案例二
DNA 甲基化在乳腺癌的发展过程中起着关键作用。之前的研究已经发现,白细胞中的 DNA 甲基化修饰可以作为一种很有前景的乳腺癌生物标记物。然而,以前的研究普遍被低统计效力和潜在的偏差限制。今年 3 月份牛津大学在 J Natl Cancer Inst(IF 11.577)上发表了一篇《Genetically Predicted Levels of DNA Methylation Biomarkers and Breast Cancer Risk: Data From 228951 Women of European Descent》, 建立了一种新的机器学习方法可以识别乳腺癌中的甲基化标志物。

研究思路
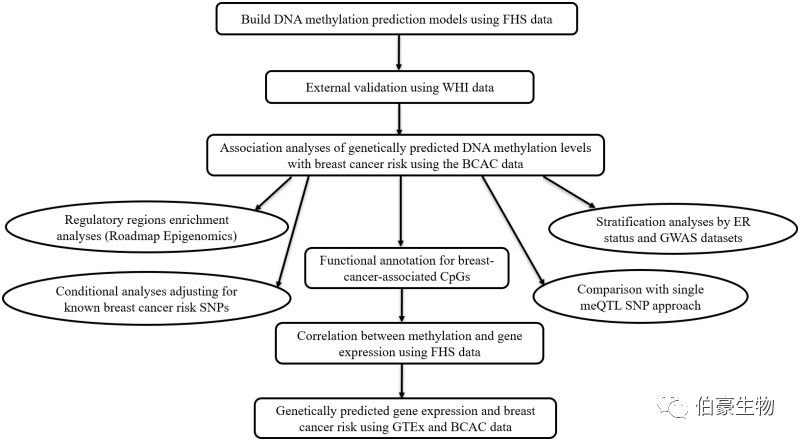
分析结果如下:
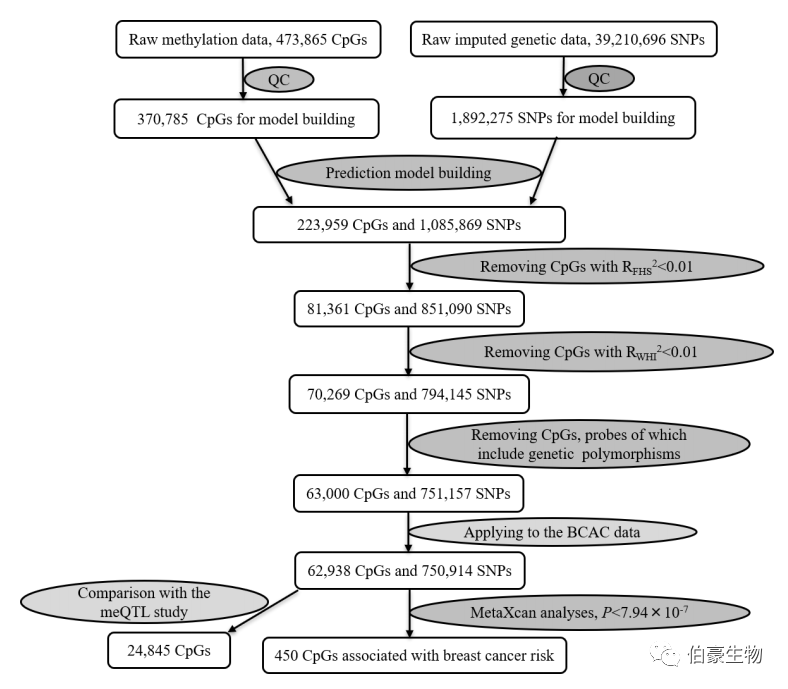
作者首先使用来自 Framingham(FHS)心脏研究中心的 1595 例患者 450K 芯片的 DNA 甲基化数据集中的多个 SNP 数据,建立统计模型来预测白细胞 DNA 甲基化标志物的水平,再使用来自 Women’s Health Initiative(WHI)的 883 患者的数据进一步对预测模型进行了验证。接着,将模型应用于 122977 例乳腺癌患者和 105974 例对照的 GWAS 数据集中,以评估所预测的 CpGs 位点的 DNA 甲基化水平是否与患乳腺癌风险相关。结果发现,在所检测的 62938 个 CpG 位点中,450 个 CpGs 位点与乳腺癌风险在统计学上显著相关,其中包括 45 个以前没有报道过的位于 18 个基因组区域的 CpGs 位点。其余 405 个 CpGs 位点位于 70 个被 GWAS 鉴定为乳腺癌风险变体的上下游 500Kb 区域内,另外 11 个 CpGs 位点独立于被 GWAS 鉴定的变异体外。 通过对 SNP 数据、DNA 甲基化和基因表达数据的综合分析,发现 38 个 CpGs 位点可能会通过调节 21 个基因的表达来影响患乳腺癌风险。 其中,GSTM4、SLC22A5 和 IMP3 3 个基因中有 5 个 CpGs 均位于 GWAS 中与乳腺癌风险不相关的基因组区域。GSTM4 过表达有助于维持细胞色素 c 的降低状态,从而增加乳腺癌细胞对甲氨蝶呤的抵抗。有报道称,SLC22A5 突变可增强乳腺组织的癌细胞转移。在 BRCA 突变的浸润性乳腺癌中发现了 IMP3 的表达增加。CD160 与细胞抗癌活性有关,有三个 CpGs 位于 CD160 中,通过下调 CD160 的表达,增加患乳腺癌的风险。另外有 3 个 CpGs 位于 MAPT 中,与乳腺癌转移有关。
(https://doi: 10.1093/jnci/djz109)
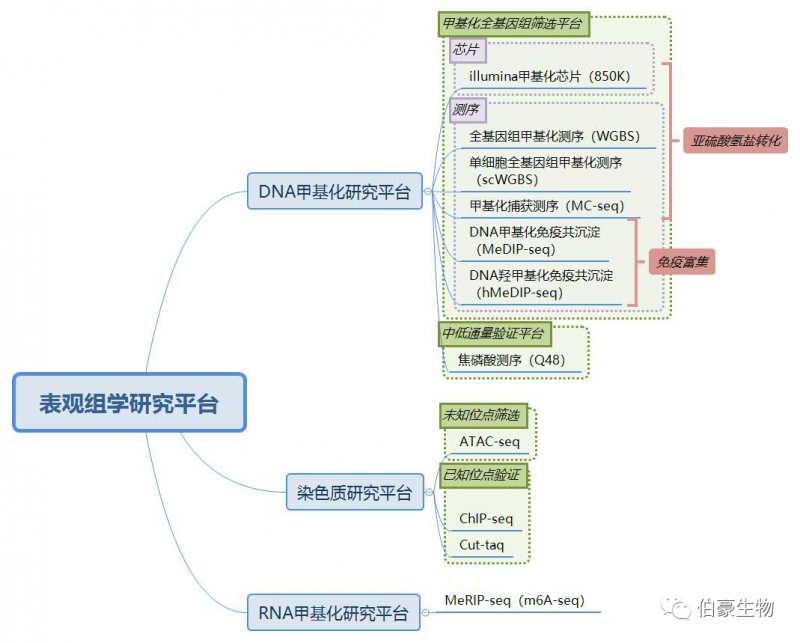
< 伯豪表观组学研究平台 >
更多伯豪生物人工服务:


