研究背景
类风湿性关节炎(RA) 的诊断主要基于临床症状和类风湿因子(RF),抗瓜氨酸肽抗体(抗 CCP) 的血清学阳性,而银屑病关节炎(PsA) 只有临床和影像学特征有助于诊断。虽然大多数 RA 患者 RF,抗 CCP 血清阳性,但约 15%-20% 的 RA 病例中,血清中 RF 和抗 CCP 水平并未升高,由于血清阴性 RA(negRA) 和 PsA 的症状非常相似,且没有可靠的临床标志物,因此 , 两者的鉴别诊断十分困难。由于治疗方法不同,可靠的诊断对于开出正确的治疗处方很重要。此外,导致 RA 和 PsA 患者特征性关节破坏的慢性炎症过程可能会引起细胞、组织和器官代谢的重大和不同程度的改变。这些代谢改变导致血清代谢物和脂质体的改变。因此研究者对 negRA 和 PsA 患者血清进行了基于 1H NMR 的代谢组学和脂质组学分析,识别 negRA 和 PsA 患者血清中浓度不同的代谢物和脂类。从这些数据中推导出一个模型,将患者分类为两种疾病类别中的一种,随后进行了盲法验证队列分析,确定生物标志物来提高这些疾病的鉴别诊断。
实验方法
检测方法:靶向代谢组学,脂质组学
检测平台:NMR
样本分组:negRA 患者 =49 例;PsA 患者 =73 例
研究结果
核磁共振波谱分析
使用 TopSpin 3.6.2 软件(Bruker BioSpin GmbH) 处理 NMR 谱。使用 AMix 软件整合单个已鉴定代谢物的相关光谱区域以进行定量。代谢物浓度的 jué对定量是通过比较来自每个代谢物的信号积分与富马酸内标的信号积分来实现的。
在 1H 单脉冲 NMR 谱中,同时观察到来自小分子和大分子的峰,导致基线不均匀和来自不同化合物的信号重叠。尽管如此,由于其特有的光谱特征,可使用 1HNMR 来识别和定量血清中的脂质。如图 1A 所示,图中有七组脂质信号(L1-L7):L1:脂质甲基;L2:脂质脂肪链;L3:脂质 β - 亚甲基;L4:脂质烯丙基亚甲基;L5:脂质 α - 亚甲基;L6:脂质多不饱和烯丙基亚甲基;L7:脂质烯烃。
在 CPMG NMR 谱中,通过抑制来自脂质和蛋白质的宽信号,所得的低分子化合物的峰不会被来自大分子的信号所掩盖,且具有清晰的信号和明确的基线,因此可以更好地识别和分析小分子产生的信号。如图 1B 所示,研究者选择了 24 中代谢物检测:(1) 甲酸,(2)组氨酸,(3)苯丙氨酸,(4)酪氨酸,(5)α- 葡萄糖,(6)脯氨酸,(7)乳酸,(8)肌酐,(9)肌酸,(10)磷酸肌酸,(11)苏氨酸,(12)胆碱,(13)肌氨酸;(14)柠檬酸盐,(15)谷氨酰胺,(16)琥珀酸盐,(17)乙酰乙酸盐,(18)谷氨酸盐,(19)乙酸盐,(20)丙氨酸,(21)β- 羟基丁酸酯,(22)缬氨酸,(23)异亮氨酸和(24)亮氨酸。
 |
图 1 PsA 和 negRA 患者的光谱特征:1H 单脉冲(A) 和 CPMG NMR(B)
多变量统计分析
对 1H 单脉冲和 CPMG NMR 光谱进行多变量统计分析,以确定是否存在可区分两种疾病的特征光谱图或峰。根据偏 zuì小二乘判别分析(PLS-DA)和随机森林模型,临床或人口统计学协变量与 1H(图 1)和 CPMG(数据未显示)光谱区域之间没有显着相关性。
 |
图 2 显示了临床 / 人口统计学变量与 1H 光谱区域之间的 Pearson 相关系数,以及用于发现队列,PsA 和 negRA 组的欧几里德度量的分层聚类
由于基于主成分分析的代谢组学数据的聚类通常很困难,用偏 zuì 小二乘法(PLS-DA) 对 1H 和 CPMG 光谱数据集进行评价。主成分 1 - 5 表明了 1H 数据方差 =96.9%/CPMG 数据方差 =84.6%,但聚类并不明显,无法清晰区分 PsA 和 negRA 患者(图 3A)。
图 3A 五个主成分之间的成对得分曲线图,对角线上显示相应的方差。
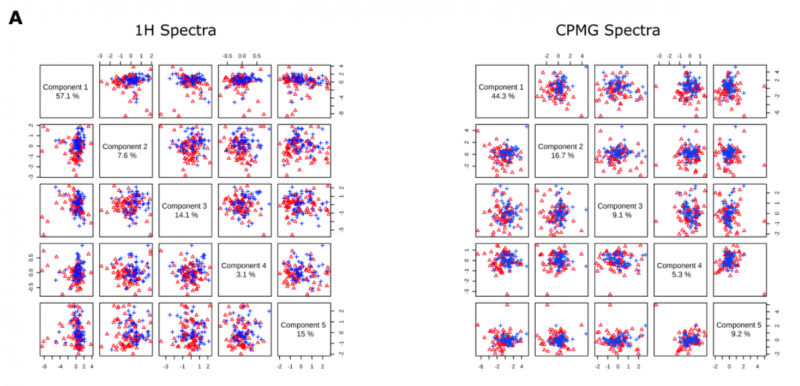 |
在评估 PLS-DA 模型的质量时,其准确度≤65%,R2 和 Q2 值都非常低(1H 光谱:R2 = 0.13,Q2 = 0.06;CPMG 光谱:R2 = 0.16,Q2 = 0.08)。为了提高基于光谱模式的诊断准确性,研究者使用了随机森林分类算法,因为它对高维数据分析具有稳健性。在 1H 和 CPMG 光谱中,该算法鉴定了对 negRA 和 PsA 患者进行分类的相似区域,基于 1H 的分类 oob error=0.361,基于 CPMG 的分类 oob error=0.336(图 3B,C)。尽管如此,通过随机森林算法确定的重要光谱区域仍被用于聚焦目标分析。
 |
图 3B 随机森林确定的显著特征。
 |
图 3C 随机森林分类的累积错误率。
(红:总体;蓝:RA;绿:PsA)
目标代谢物,脂类聚焦分析
在定量代谢物和脂质组的浓度后,通过单变量分析确定患者组之间的差异。在 24 种代谢物中,两种患者组中的 9 种具有明显不同的浓度,即氨基酸(AA):丙氨酸,亮氨酸,苯丙氨酸,苏氨酸和缬氨酸以及有机化合物乙酸盐,胆碱,肌酸和乳酸。
另外发现 negRA 和 PsA 患者之间的脂质比率 L3 / L1,L5 / L1 和 L6 / L1 有统计学差异在 PsA 中均较高(图 4A)。比较两组时,某些代谢途径明显富集(图 4B)。
 |
图 4(A)目标分析中包括的代谢物和脂质比率的点状图,这些点状图显示了发现队列中两个患者组之间的显着差异。线表示平均值和 95%CI。(B)定量富集分析的摘要条形图,显示发现队列中 negRA 和 PsA 代谢组之间的变化。
多元方差分析(MANOVA)
年龄,性别和治疗方案可能会影响不同疾病中生物体液中代谢物的浓度,从而影响要用于初治患者或不同年龄患者的生物标志物的定义。为了分析任何临床或人口统计学参数是否可能对 24 种代谢物或脂质组的血清浓度产生影响,研究者对相关代谢物和潜在代谢物进行了多元方差分析(MANOVA)。
结果表明(表格未展示)疾病活动与胆碱浓度,L2 / L1 和 L7 / L1 的变化有关,而疾病持续时间与柠檬酸盐,磷酸肌酸,葡萄糖,组氨酸,酪氨酸和缬氨酸浓度的变化有关。当将年龄和体重指数类别与疾病组结合时,代谢物浓度和脂质比率的变化同样可见。尽管 RA 是一种主要影响女性的疾病,与 PsA 相反,但 MANOVA 分析结合了疾病类别和性别,在相关代谢产物上没有任何显着差异。当疾病和治疗相结合时,情况类似。单变量分析显示代谢物浓度或脂质比率与临床和人口统计学变量之间有没有任何显著相关。如图 5
 |
图 5 显示临床或人口统计学变量与代谢物之间的 Pearson 相关系数,并使用欧几里德度量对发现队列的 PsA 和 negRA 组进行分层聚类。
用于患者分类的多元诊断模型
单一代谢物或脂质比例的 ROC 分析得到的曲线下面积(AUC) 小 70%。因此,单变量模型对 PsA 和 negRA 患者的鉴别的敏感性和特异性低。为了达到高的诊断准确率,研究者建立了三种不同的机器学习算法:随机森林算法、朴素贝叶斯算法和多元 Logistic 回归算法,对 73 例 PsA 和 49 例 negRA 患者的代谢组和脂质组进行了分析。随机森林预测 PsA 的准确率 73.3%(Cohen‘s kappa 40.1%),朴素贝叶斯准确率为 63.7%(Cohen’s kappa 26.5%)。
通过逐步向前 - 向后选择算法,以下诊断预测因子已包括在诊断模型中:年龄,性别,L6 / L1,L5 / L1,L2 / L1,丙氨酸,琥珀酸和磷酸肌酸。
在验证过程中,使用十折交叉验证对所得到的模型进行评估,这产生了表 1 中的系数估计。
 |
表 1 模型系数的评估
将这些估计值应用到回归模型中会产生以下公式:
 |
每种代谢物的浓度、年龄和性别(男性 =1,女性 =0) 代入公式中。然后,通过替换在 F1 中获得的结果×来计算属于 PsA 组的概率:

患者属于 negRA 组的概率由下式给出:

将患者分为两组,需计算概率(F2) 的分界值。通常应用分界值 0.5,PPsA>0.5,则将其分类为 PsA,这反映了将对象分类为更有可能进行诊断的想法。图 6 为发现队列中交叉验证的建模概率 PPsA 的 ROC 曲线,该图显示了该模型对此临界值的敏感性和特异性,ROC 曲线下的总面积(AUC) 为 84.5%。
为了进一步验证从交叉验证过程中检索到的模型,研究者收集了 35 名新的关节炎患者的单独的盲法样本。该队列与用于建立诊断模型的队列具有相似的路径分布(图 7)。使用临界值 0.5 对盲法队列进行评估,得出了 62.9% 的患者的正确预测,并进行了 ROC 分析,其中 AUC 降至 71.6%(图 8)。
在诊断预测后,对 13 名预测诊断与风湿科医生 zuì 初所做的诊断不同的患者进行了临床重新评估。对于这些患者中的 4 名,仍然无法确定诊断,因为他们一直缺乏独特的临床参数。研究者从验证队列中删除了这四个个体,重新计算了预测匹配率,增加到 71.0%,并进行了新的 ROC 分析,结果提高了敏感度(62.5%) 和特异度(80.0%)(图 9)。
|
▲图 6
 |
 |
▲ 图 8
 |
结论
为验证其在基因异源人群中的有效性,还需要在更大的多国 / 多民族队列中进行测试。尽管如此,研究者的研究有助于将 1HNMR 的代谢组学这一技术扩展到自身免疫病理学的常规诊断技术铺平道路。即使临床特征相似,与风湿性疾病相关的代谢组学过程在疾病之间也有所不同。因此,代谢组学和脂质组学开始为自身免疫研究提供一个全新的领域。
参考文献
Margarida souto-carneiro,lilla Tóth,rouven Behnisch,et al. Differences in the serum metabolome and lipidome identify potential biomarkers for seronegative rheumatoid arthritis versus psoriatic arthritis. Ann Rheum Dis.2020; 79(4):499-506.
更多伯豪生物服务



