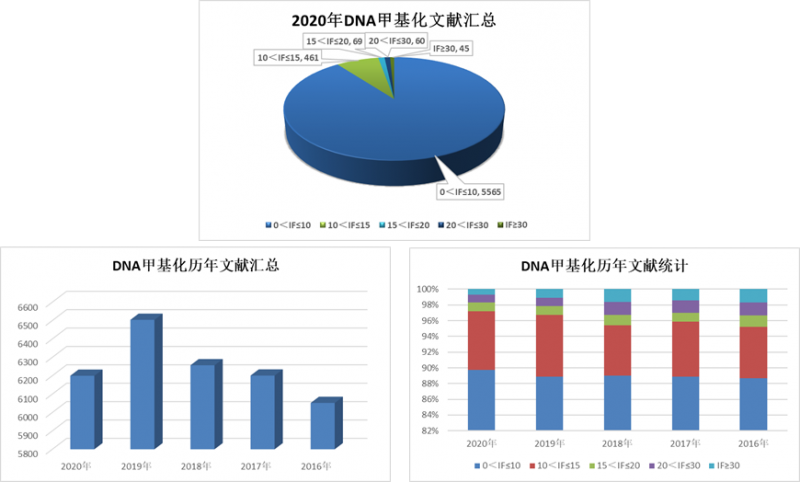
在刚刚过去的 2020 年中,以 DNA methylation 为关键词在 Pubmed 中共能检索到 6200 篇文章。其中,IF>10 的文章共计 635 篇,IF>20 的共计 105 篇,IF>30 的共计 45 篇。和前五年呈现出的显著递增趋势相比,2020 年所发表文献的数量有明显下降,高分文献的占比有所减少,这和去年的新冠疫情影响应该有很大关系。
虽然如此,2020 年 DNA 甲基化研究的高分文献也有很多值得研读的,小编也在这里精挑细选了几篇不同研究思路的高分甲基化文章(IF>20)和大家重点分享。
一、DNA 甲基化多中心研究(中介效应研究)
2020 年 2 月 18 日,比利时天主教鲁汶大学医院、荷兰鹿特丹伊拉斯谟大学医学中心索菲亚儿童医院和加拿大阿尔伯塔大学斯托雷儿童医院在《Lancet Respir Med》(IF 25.094)联合发表“Effect of early parenteral nutrition during paediatric critical illness on DNA methylation as a potential mediator of impaired neurocognitive development: a pre-planned secondary analysis of the PEPaNIC international randomised controlled trial”——“PIUC(儿童重症监护病房)患者早期 PN(早期补充肠外营养)以 DNA 甲基化为潜在介质对神经认知发育受损的影响”。

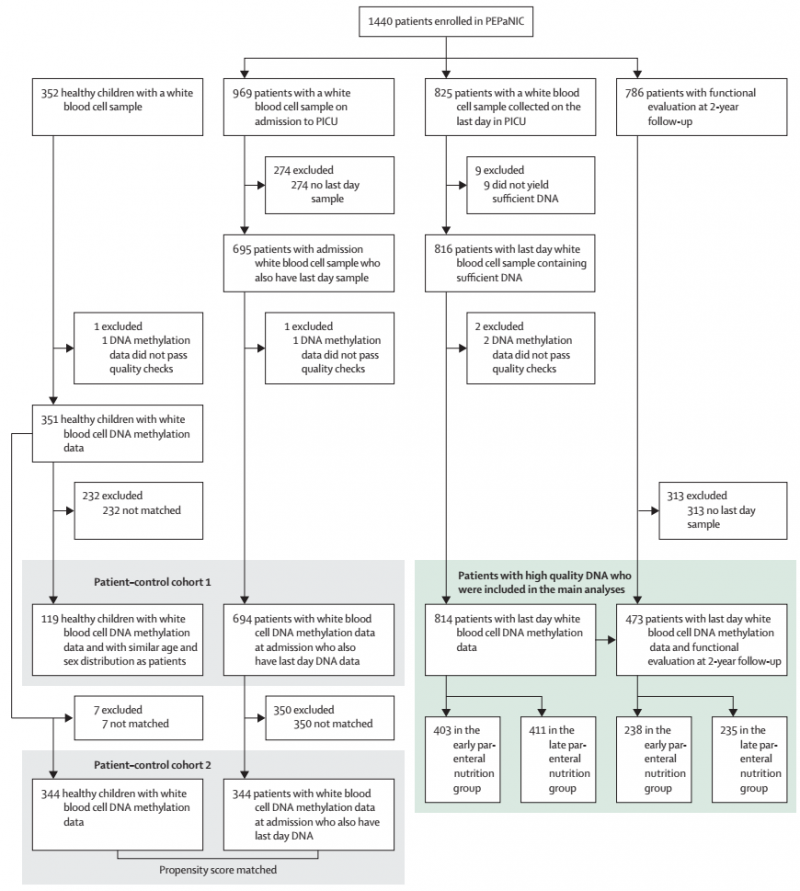
作者在 17 年同篇发表的文章中,通过大型随机对照实验 PEPaNIC 发现早期 PN 会对儿童神经认知发育产生负面影响。这项针对多中心 PEPaNIC 试验(2012-2018)的分析主要包括收集到在 PICU 后一天患者的血液样本(n = 825,PICU 入院时年龄为 0 -17 岁),其中早期 PN 和晚期 PN 的患者各占一半,并将这些样本与相匹配的 352 位健康儿童进行比较。结果发现和健康儿童相比,PICU 住院患者存在 159 个差异 CpG 位点甲基化位点,这些差异甲基化的 CpG 位点主要位于大脑发育,可塑性和信号传导等相关的基因上。同时,作者使用多元线性和非线性回归分析来评估早期 PN 与晚期 PN 对疾病引起的 DNA 甲基化改变的影响,以及 CpG 位点甲基化水平可以在多大程度上解释 2 年后神经认知发育受损的程度。结果发现,早期 PN 中的氨基酸剂量会导致其中 37 个差异 CpG 位点的甲基化差异,说明早期 PN 中的氨基酸剂量是对影响神经认知发育的主要因素。
参考文献 DOI: 10.1016/S2213-2600(20)30046-1
二、癌症 ctDNA 甲基化标志物研究
循环肿瘤 DNA (Circulating tumor DNA, ctDNA) 是肿瘤细胞破裂掉落下来的 DNA 片段,进入了外周血循环系统。ctDNA 携带了肿瘤基因组一定的突变信息,还携带甲基化 5mC/ 羟甲基化 5hmC 等信息,在肿瘤早筛早诊等方面具有独特的应用价值,是液体活检中的重要分支。
中山大学肿瘤防治中心徐瑞华教授课题组在《Sci Transl Med》(IF 16.304)上发表“Circulating tumor DNA methylation profiles enable early diagnosis, prognosis prediction, and screening for colorectal cancer”——“应用 ctDNA 甲基化标志物可进行结直肠癌(CRC)的早期诊断,预后预测和筛查”,这是继 17 年发表在《Nature materials》(IF 38.663)肝癌 ctDNA 甲基化标志物研究后的又一篇佳作。

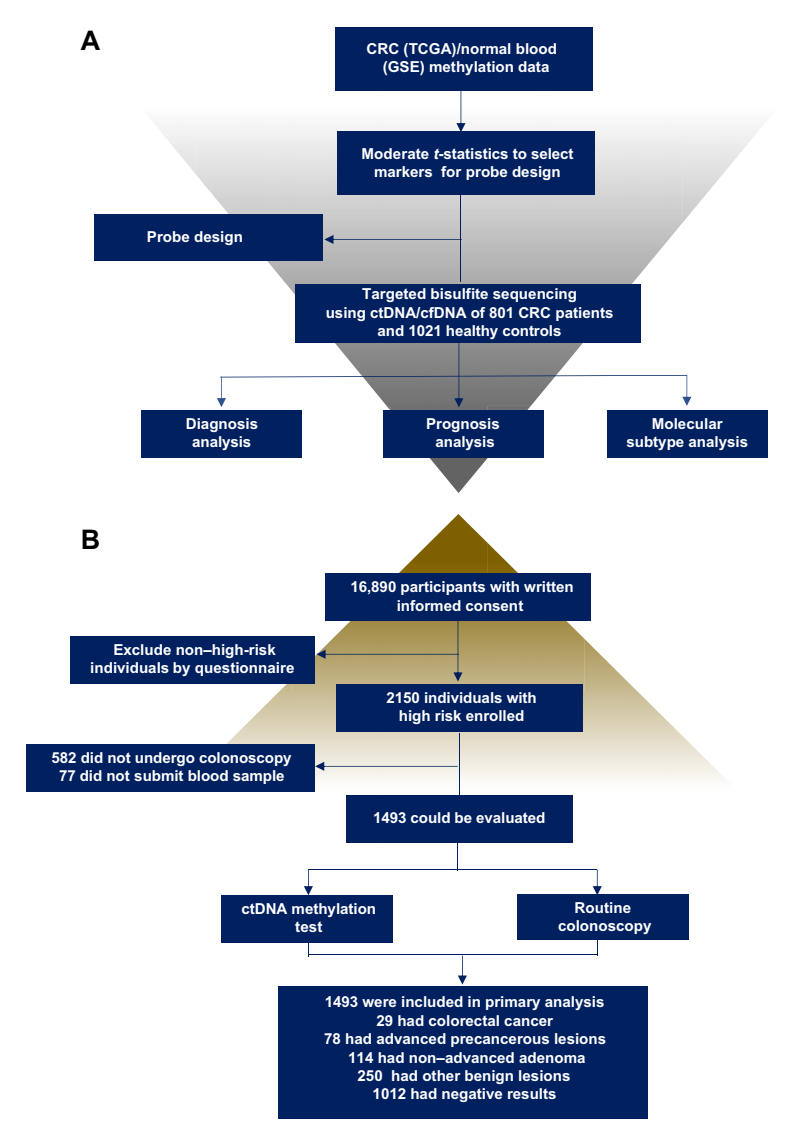
作者首先基于公共数据库筛选到 544 个 CpG 位点,并利用探针捕获测序的方法在 801 例 CRC 患者 /1021 例健康人群血浆中提取的 cfDNA 进行了训练,筛选到 9 个 CpG 位点构建诊断模型,训练集和验证集中该模型诊断率高度一致(AUC=0.96),且准确性明显高于目前临床上使用的肠癌血检标志物——癌胚抗原(CEA)(AUC=0.67)。同时,作者筛选到 5 个 CpG 位点用于构建预后模型,将得到的预后评分指数(cp-score)联合目前常用的临床预后指标,如肿瘤原发部位、TMN 分期、CEA 等,能明显增强预后预测的准确性。接着,作者分别分析了诊断模型中的 9 个 CpG 位点,其中 cg10673833 位点的 AUC 为 0.91,在前瞻性队列(n=1493)中验证该位点的对肿瘤的检出敏感性达 89.7%,特异性达 86.8%,对进展期腺瘤的检出率敏感性达 33.3%,敏感性和特异性均较现有的无创筛查方法有所提高。
参考文献 DOI: 10.1126/scitranslmed.aax7533
2020 年 06 月 12 日,《Nature Medicine》(IF 36.13)同期发表了两篇文章使用 cfMeDIP-seq 技术分别寻找肾细胞癌和颅内肿瘤 ctDNA 甲基化标志物的文章,小编就不再这里再进行一一介绍了,感兴趣的小伙伴可以通过下边的 DIO 号获取或者联系我们直接获得全文哦~
参考文献:
Nuzzo P V , Berchuck J E , Korthauer K , et al. Detection of renal cell carcinoma using plasma and urine cell-free DNA methylomes[J]. Nature medicine, 2020, 26(7).
Nassiri F , Chakravarthy A , Feng S , et al. Detection and discrimination of intracranial tumors using plasma cell-free DNA methylomes[J]. Nature medicine, 2020, 26(7).
三、DNA 甲基化多组学研究
2020 年 3 月 25 日,海军军医大学长海医院孙颖浩院士领衔在《Nature》(IF 42.778)发表“A genomic and epigenomic atlas of prostate cancer in Asian populations”,揭示了中国前列腺癌人群特有的多组学分子特征。

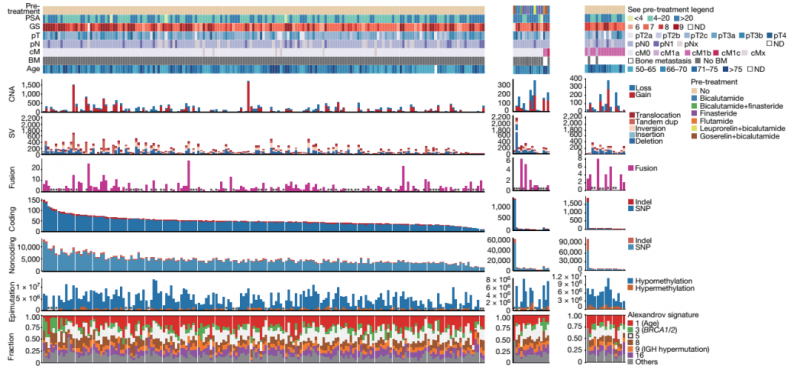
作者通过对 208 例原发性前列腺癌的癌组织与癌旁组织进行 WGS、WGBS、RNA-seq 和 miRNA-seq 检测,分别从 CAN、SV、融合基因、编码区和非编码区的 SNV/Indel、胚系表观突变多个水平展现中国人群前列腺癌基因组和表观基因组图谱(CPGEA)。同时,在此基础上,作者将该数据与 2554 例西方患者比较发现,中国患者的基因组突变与西方患者存在明显差别,这为中国前列腺癌人群的个性化治疗提供了理论依据。
参考文献 DOI: 10.1038/s41586-020-2135-x
2020 年 7 月 13 日,密歇根大学在《Nat Genet》(IF 27.603)发表了关于前列腺转移癌的全基因组与表观基因组图谱的研究成果——“The DNA methylation landscape of advanced prostate cancer”,该项目使用了西方人群的大队列进行研究,感兴趣的小伙伴可以联系我们即可轻松获取文献哦~
参考文献:
Gagliardi A , Porter V L , Zong Z , et al. Analysis of Ugandan cervical carcinomas identifies human papillomavirus clade–specific epigenome and transcriptome landscapes[J]. Nature Genetics.
Zhao S G , Chen W S , Li H , et al. The DNA methylation landscape of advanced prostate cancer[J]. Nature Genetics, 2020, 52(8).
Carrillo-Reixach J , Torrens L , Simon-Coma M , et al. Epigenetic footprint enables molecular risk stratification of hepatoblastoma with clinical implications - ScienceDirect[J]. Journal of Hepatology, 2020, 73(2):328-341.
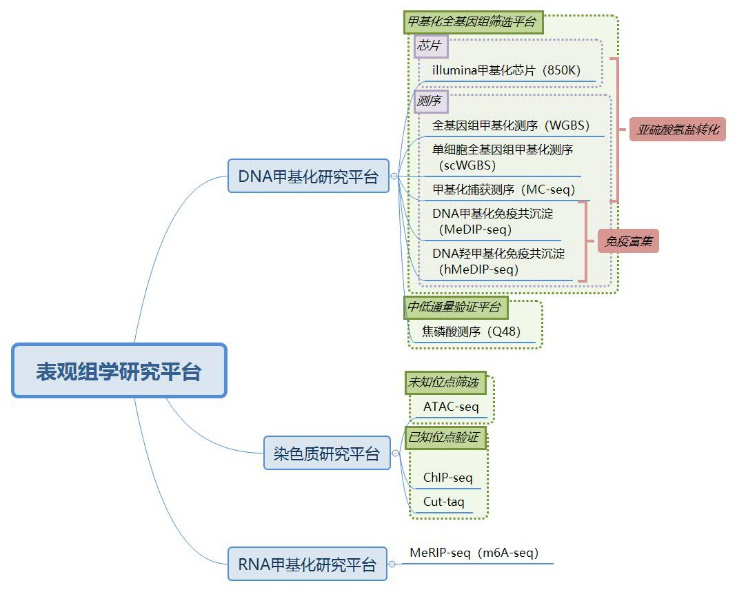
伯豪生物甲基化研究平台:
更多伯豪生物人工服务:


